Metabarcoding de l'ADN environnemental et des méthodes traditionnelles d'inventaire
Comparaison de l’utilisation metabarcoding de l'ADN environnemental et des méthodes traditionnelles d'inventaire pour l'évaluation de la biodiversité des mammifères
Les mammifères, composants vitaux des écosystèmes terrestres, font face à des déclins drastiques en raison des changements anthropogènes dans les paysages. Avec de nombreuses espèces isolées dans des zones protégées et confrontées à un statut de conservation extrême, il est de plus en plus nécessaire de disposer de méthodes rapides, rentables et non-invasives pour surveiller leurs populations. Au sein de BCOMING, il est important que nous utilisions les techniques les plus précises pour établir l'évaluation de la biodiversité, car elle est une composante essentielle de nos recherches sur la circulation des agents pathogènes. Notre équipe de l'Université de Liège en Belgique, composée de la Dr Pauline van Leeuwen et du Prof. Johan Michaux, s’est plongée dans la littérature scientifique à la recherche de méthodes non-invasives pour détecter les espèces de mammifères sur le terrain. Ils ont publié le premier article scientifique soutenu par le consortium BCOMING. Cette étude compare les méthodes d'inventaire conventionnelles et la technique émergente de metabarcoding de l'ADN environnemental (eADN) pour l'évaluation de la biodiversité des mammifères.
L'essor du metabarcoding de l'ADN environnemental: Les méthodes d'inventaire conventionnelles, y compris le piégeage et les enquêtes sur le terrain, ont été la pierre angulaire de l'évaluation de la biodiversité des mammifères. Cependant, l'étude met en avant l'émergence du metabarcoding de l'ADN environnemental comme un outil de détection de plusieurs espèces basé sur les technologies de PCR et de séquençage de nouvelle génération (NGS). Cette technique utilise l'ADN extrait d'échantillons environnementaux, offrant un moyen non-invasif de surveiller la biodiversité. Les échantillons d'eADN peuvent être catégorisés en trois groupes en fonction des substrats : l'eADN de l'environnement (eau, sol, sédiments et filtres à air), les pièges à ADN (fèces, poils, salive, végétation, et même toiles d'araignées !), et l'ADN dérivé d'invertébrés (sangsues, mouches, moustiques, scarabées coprophages). Chaque substrat offre des avantages uniques pour capturer le matériel génétique, mettant en valeur la polyvalence du metabarcoding de l'eADN.
Avantages et défis du metabarcoding de l'eADN: Comparé aux méthodes traditionnelles, le metabarcoding de l'eADN présente plusieurs avantages, notamment une main-d'œuvre réduite, une collecte standardisée dans tous les habitats et la non-invasivité. Cependant, des défis tels que la dégradation de l'ADN, les erreurs de PCR et la nécessité de vastes bases de données de référence doivent être abordés. L'étude souligne l'importance d'une approche nuancée et de la validation croisée avec les méthodes traditionnelles.
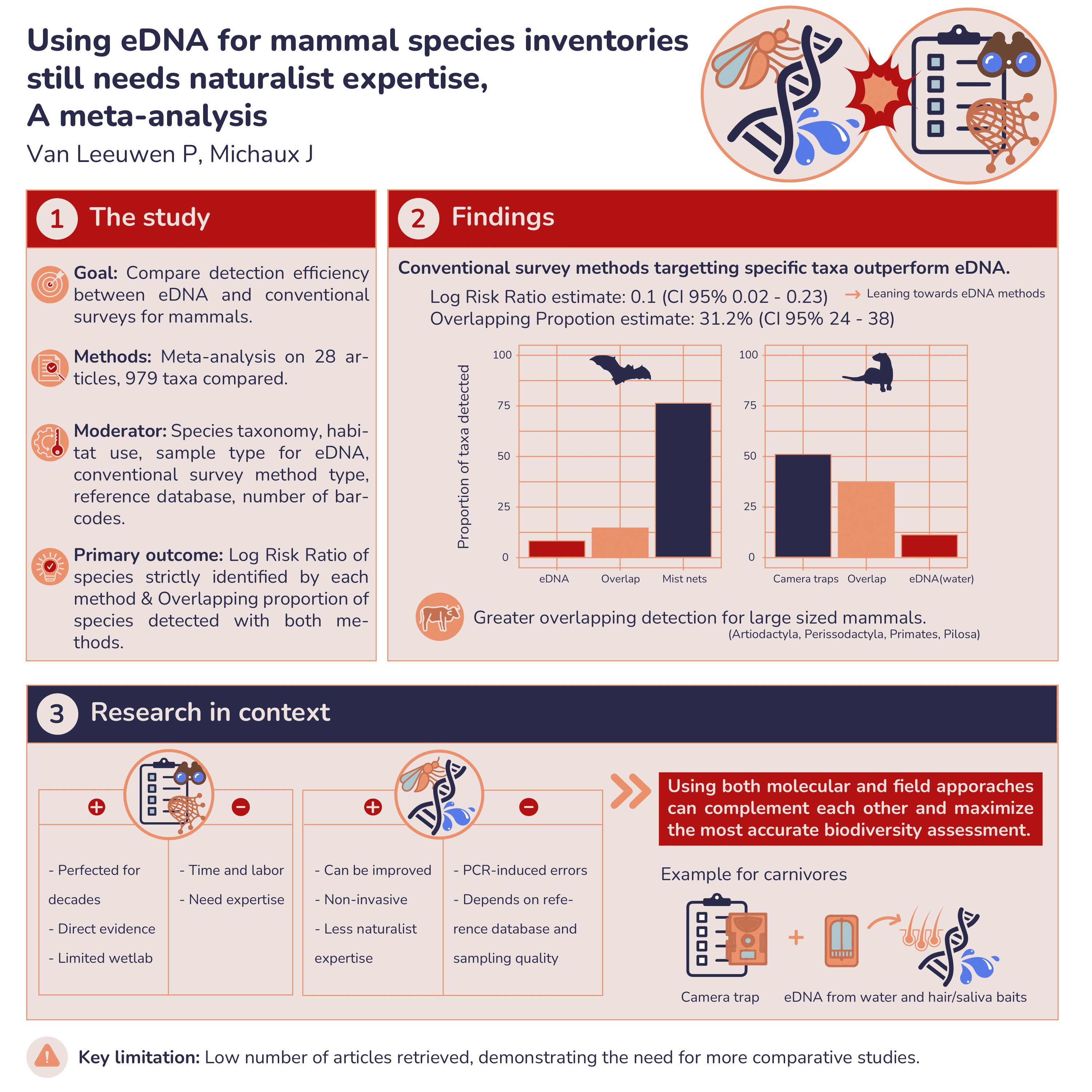
Succès de la détection dépendant de la taxonomie des espèces recherchées: Une méta-analyse exhaustive, couvrant la période de 2005 à 2023, révèle des informations sur le chevauchement de la détection entre le metabarcoding de l'eADN et les méthodes traditionnelles. La taxonomie, l'utilisation de l'habitat, le type d'échantillon, les méthodes d'inventaire traditionnelles, les bases de données de référence et le nombre de codes-barres utilisés sont identifiés comme des modérateurs influençant le succès de la détection. Une méta-régression affine davantage l'analyse, fournissant une compréhension nuancée des facteurs influençant les taux de détection.
Notre étude explore le succès dépendant de la taxonomie du metabarcoding de l'eADN, indiquant des variations dans l'efficacité de la détection à travers les ordres de mammifères. Les carnivores présentent un plus grand succès de détection avec les méthodes traditionnelles, en particulier les pièges photographiques, tandis que l'eADN fonctionne bien pour les ongulés et les primates. L'étude souligne la nécessité d'approches adaptées en fonction des caractéristiques physiologiques et écologiques des espèces cibles.
Dans l'ensemble, nous démontrons le potentiel prometteur du metabarcoding de l'eADN pour l'évaluation de la biodiversité des mammifères. Cependant, nous soulignons également la nécessité d'une approche hybride, combinant les forces de l'eADN et des méthodes d'inventaire traditionnelles. Reconnaissant la nature dépendante de la taxonomie du succès de la détection, nous appelons à une intégration réfléchie des outils moléculaires et de l'expertise sur le terrain pour maximiser la précision des évaluations de la biodiversité. Alors que l'eADN continue d'évoluer, des efforts collaboratifs entre les biologistes moléculaires, les taxonomistes et les écologistes sur le terrain sont cruciaux pour affiner les méthodologies et élargir notre compréhension des populations de mammifères.
Pour plus d'informations, voici le lien en accès libre vers la publication: https://doi.org/10.1002/ece3.10788